4A compare and contrast prokaryote and eukaryote cells Cells.
L2 Prokaryote vs Eukaryote Cells Archaea · 2018-02-23 · L2 Prokaryote vs Eukaryote Cells...
Transcript of L2 Prokaryote vs Eukaryote Cells Archaea · 2018-02-23 · L2 Prokaryote vs Eukaryote Cells...

L2 Prokaryote vs Eukaryote Cells
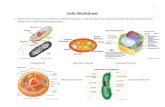
Prokaryotic Cells: (pro: before, karyon: nucleus) are cells in which the double stranded DNA lies free within the cell (in an area ‐ the nucleoid)
Ribosomes can attach directly to mRNA, even while being synthesised in the cytoplasm
Eukaryotic Cells: (eu: proper, karyon: nucleus) are more complex cells that arose from, bacterial ancestors. DNA is enclosed within a nucleus
Prokaryotic Cells Have a smaller in simpler structure that eukaryotes
Prokaryotes are the smallest cellular like on earth
and differ structurally and functionally from eukaryotes.
They are Earth's first inhabitants (dating back ~3.6 billion years), thus have evolved with high diversity, and have adapted to almost every climate
CYANOBACTERIA (photosynthetic) produces O2 for the previously oxygen less atmosphere (The Great Oxygenation Event)
► Caused the extinction of many anaerobic organisms
► The first billions of years saw no change ‐> O2 bound to iron
► O2 was split by the radiation of the sun ‐> created the ozone layer
Produce resting spores that can lay dormant for >250 million years
Split every 20 mins (BINARY FISSION)
Three Domains of Life The bacteria
The Archaea
The Eukarya
Archaea (very old life forms)
Morphologically identical to bacteria (no internal compartments
Over half of Archaean genes were new to science ► Discovered by the testing of nucleic acid
Biochemically, Archaea are nearly as different from bacteria as they are from Eukarya
► Making them a separate domain ► Archaean transcription & translation are
more similar to Eukaryotes
Lack a peptidoglycan wall
Most were believed to be EXTREMOPHILES ‐ found in extreme environments
None have been found to produce resting spores
No clear examples of Archaean pathogens known
Comparison of Structural + Biochemical Features
Feature Bacteria Archaea Eukarya
Membrane‐bound nucleus
absent absent Present
Introns in genes absent absent Present
Plasmids present present Rare
RNA polymers single multiple Multiple
Protein synthesis ‐initiation RNA
Formyl methionine
Methionine Methionine
Peptidoglycan Yes No No
Membrane lipids Esters Ethers Esters
Methane‐generating No Yes (some) No
Nitrification Yes (some) No No
Nitrogen fixing Yes (some) Yes (some) No
Photosynthesis Yes (some) No Yes (some)
Protein synthesis sensitive to:
Diphtheria toxin
Streptomycin Cycloheximide
No Yes No
Yes No No
Yes No No
Prokaryotes Functions and Features Ubiquitous and metabolically diverse
Cause of many diseases
Decomposers and recyclers ► Removes dead organic matter, recycling
carbon, nitrogen in water to safe levels
Agents in industrial and agricultural processes ► In fermented foods e.g. salami, soy sauce
Nitrogen fixation ► Fixes atmospheric (unusable) nitrogen to
organic compounds ‐ can then be accessed by other organisms
Biotech applications ► GM bacteria make pharmaceuticals e.g.
harvesting pathogen free hormones ► GM plants

Cyanobacteria ‐ major primary producer
Contain chlorophyll A, plus phycocyanin and phycoerythin as accessory pigments
Resemble algae and plants
Eukaryotes have almost equal numbers of genes from both Archaeans and Bacteria
Possess internal, membrane‐bound organelles
Has a semi‐permeable cellular membrane
L3 Eukaryotic Cell
The Nucleus Surrounded by the nuclear envelope
► Double membrane ► Separated by 50um, but joined at the pores
~30 proteins surround each pore acting as filters ► Substances need proteins (NLS, NES) to
enter and exit the pores
DNA in the nucleolus are covered with histones to form chromosomes
► Histones are used to compact the DNA ► They are +vely charged and balance out the ‐
ve charge of DNA
RNA transcribes from DNA leaves the nucleus via pores and is translated in the cytoplasm
The nucleolus contains rRNA, proteins, DNA and ribosomes in various stages of synthesis.
Mitochondria Cellular respiration occurs in the mitochondria in all
eukaryotes
Cells may contain several, or have a single large one
They have two membranes ► An outer membrane ‐ highly
permeable ► And a highly convoluted inner
membrane ‐ highly permeable
Chloroplasts Have:
► An outer membrane ► A complex internal network of lamellae or
thylakoids Form stacks called grana Where pigments are and
photosynthetic reactions take place
Stroma ‐ liquid surrounding thylakoids (high pH)
Lumen ‐ liquid within thylakoids (low pH)
Accessory pigments ‐ captures other wavelengths (photons) that chlorophyll A might miss, then passes it to chlorophyll A
► Makes the organism appear in various shades of brown or red
Complex Organelles Derived from Relict
Symbionts (Endosymbiont) An organism living (operating) inside another
organism where they both benefit each other ► MITOCHONDRIA are believed to be derived
from PURPLE BACTERIA ► CHLOROPLASTS are believed to be derived
from CYANOBACTERIA
Primary Endosymbiosis (plastids with two
membranes)
99% of the time, objects absorbed my phagocytosis become dissolved
There may have been a mutation in the cyanobacteria cell wall that made it unrecognisable as food
Over time, the cyanobacteria became a plastid ► Lost its autonomy ‐ controlled by the nucleus ► Food vacuole disappears and becomes a
chloroplast
Secondary / Eukaryotic Endosymbiosis When a chloroplast is believed to be derived from a
symbiotic, eukaryotic cell rather than a prokaryote.

Protists: have chloroplasts taken from other eukaryotes ► Single celled ► Produce ~60% of the world's oxygen
Evidence for the Endosymbiotic origin 1. These organelles appear morphologically similar to
bacteria 2. They are surrounded by a membrane similar to a
cell membrane, while their inner membrane invaginates to form thylakoids / cristae
3. Mitochondria & chloroplasts are semi‐autonomous ► Retains their own genome
4. They also retain their own machinery for synthesising proteins, including ribosomes
5. Their metabolism are like existing prokaryotic organisms (CYANOBACTERA/PURPLE BACTERIA)
6. Some chloroplast still have the bacterial peptidoglycan wall between inner and outer membranes (e.g. cyanophora)
► Evidence that it was a domain bacteria 7. Chloroplast metabolism is like that of existing
prokaryotic cyanobacteria CRYPTOMONADS: flagellates with secondary plastids (has 4 genomes)
Cryptomonad plastids have chlorophyll and a phycobilin pigment
► Products of photosynthesis is stored outside the plastid as starch
Reproduce asexually, are unicellular
Have a nucleomorph, a second vestigial nucleus ► Proof that the endosymbiont was a
photosynthetic eukaryote
Mitochondria & chloroplasts contain genetic material themselves
Ribosomes, cytoplasm, DNA. Plasma membrane; are features that prokaryotes and eukaryotes share.

L4 The Endomembrane System A system of membrane‐bound
compartments( including nuclear envelope) ► Excluding the mitochondria and chloroplasts
THE FUNCTION
Provide a surface for biochemical reactions
Establish compartments to prevent mixing
Provide for transport of materials
They always enclose a space ‐ unless damaged
They are always rounded ‐ never form T‐junctions
Membranes are the consistency of olive oil
Endoplasmic Reticulum Consists of membrane cisternae (stacks) that
branch out through the cytoplasm ► Usually flat and sheet‐like, but are often
linked by tubular cisternae ► Resulting in internal compartments and
channels
It is attached to the outer membrane of the nuclear envelope (functionally and physically)
50% of the cell's membrane volume is ER
The ER is a dynamic structure, ever changing in structure and function
ROUGH ER: has ribosomes attached throughout ► Provides surface3s for protein synthesis
SMOOTH ER: do not have ribosomes attached ► Provides surfaces for lipid and
carbohydrate synthesis
Golgi Apparatus Consists of flattened stacks of membrane/cisternae
Golgi stacks are functional extensions of the ER
They are polar structures ► Have a forming face and mature face where
vesicles enter and exit
Collects, packages and distributes molecules synthesised from elsewhere
The polysaccharide may be attached to either protein or lipid molecules
► Evolving into their mature form
Proteins leave the cell in secretory vesicles from the Golgi body
Lysosomes Only appear in animal cells, surrounded by a single
membrane
Break down worn‐out cells via autophagy ► Macromolecules break down into protein,
carbohydrates, lipids and nucleic acid ► Reused or secreted
Break down material ingested from outside via endocytosis/phagocytosis
Has an acidic interior and ~40 different hydrolytic enzymes
► Have a highly specialised membrane
Plant Vacuoles He plant equivalent of lysosomes, surrounded by a
single membrane (tonoplast)
Contain hydrolytic enzymes and serve as degradative compartments
It also stores nutrient, pigments, waste materials, and maintains cell turgor pressure
Micro bodies Main organelle of removing compounds generated
within the cell, surrounded by a single membrane
Neutral pH; contain oxidative enzymes that generate H2O2 and the enzyme catalase to break down the H2O2
There are two types: PEROXISOMES: oxidise amino acids and uric acid
GLYCOXYSOMES: convert fatty acids to sugars
The Cytoskeleton Composed of proteins, NOT membranes
Proteinaceous compounds
Cytoskeletal components act as structural elements within the cytoplasm and fix organelles in position
Also are associated with maintaining cell shape, and involved in certain cell movements
MICROTUBULE: constructed of alternating α and β tubulin protein
► Polar, embeds ‐ve end, grows from +ve end ACTIN (MICRO)FILAMENT: composed of actin ‐ monomer of repeating protein
► The smallest INTERMEDIATE FILAMENT: not dynamic (medium sized)
Form stiff structures that don't branch or contract
Are polar and highly dynamic
Polymerise and depolymerise to satisfy cell needs
Cell movements are generated by "motor proteins" associated with actin filaments and microtubules
Microtubule Associated Proteins (MAPs)
KINESIN motors: move towards the +ve end of the microtubule DYNEIN motors: move towards the ‐ve end, motors move by ATP hydrolysis
MAP dynein motors cause sliding of adjacent microtubule doublets relative to each other and generate flagellar movement
MYOSIN motors: pull organelles along actin filaments
Responsible for cytoplasmic streaming as well as muscle contraction

Cytoplasmic Streaming (Plant) Chloroplasts may or may not stream
Moves nucleus around cell
Plant Cell Wall Composed primarily of cellulose
► Polysaccharide of linked D‐glucose units
Most abundant organic molecule on earth
Future biofuel ‐ cellulosic ethanol
L5 Cell Division Cell division is the process whereby one cell divides to generate two new cells.
The cytoplasm and identical copies of its DNA are transmitted to each daughter cell when a cell divides
The rate and number of cell divisions in multicellular organisms must be regulated for tissues to form correctly and be the right size
► Abnormal development and diseases (cancer) result from changes in cell cycle regulation.
Required for growth, replacement for aged cells, for reproduction
Mitosis: DNA replication Cytokinesis: division of the actual cell
Prokaryote Cell Division Prokaryotic cell division is simple, and involves replication of a single circular strand of DNA, followed by simple cell division by binary fission.
Eukaryotic Cell Division DNA replication and nuclear division occurs prior to cytokinesis
Cell Cycle Interphase: during which DNA and most of the other molecules required by the cell are synthesised. Cells progressing through interphase to the next division only show subtle changes in their appearance because DNA exists as de‐condensed chromatin (individual chromosomes are not visible)
G1 PHASE: the phase which immediately follows the completion of mitosis ► the first and longest part ► cell grows in preparation for cell division ► normal metabolism, duplication of
organelles
S PHASE: the period during which DNA is replicated
► at the end of s phase, the nucleus is appreciably larger and contains twice the amount of DNA
► Each chromosome is duplicated. ► sister chromatids are held together by a
centromere and are not considered separate chromosomes until they separate
G2 PHASE: period of cell size increase in prep for cell division, final preparations for mitosis occur
► Checks that replication has been faithfully completed.
► Once a cell enters M PHASE, it cannot stop or go back. It must continue until mitosis is completed.
M PHASE Prophase: chromatin in the nucleus condenses into chromosomes; mitotic spindle begins to form; centrosomes duplicate and move apart Prometaphase: nuclear envelope breaks down Metaphase: chromosomes line up at the middle of the spindle (metaphase plate) Early anaphase: sister chromatids separate and start moving towards the poles Anaphase: individual arms of chromosomes can be seen as chromosomes move towards the poles Early telophase: the chromosomes begin to de‐condense and cytokinesis has commenced Telophase: the nuclear envelope has reformed CYTOKINESIS: begins in late mitosis
involves the pinching of the cytoplasmic membrane to create two new cells
this constriction is brought about by a contractile ring
PLANT CELL CYTOKINESIS
fibres remaining between the chromosomes thicken and accumulate into a densely fibrous phragmoplast
this steadily grows out laterally until it reaches the older walls of the cell
inside, membrane vesicles appear and slide along the fibres, collecting halfway between the reforming nuclei and fusing to form a new sheet of cytoplasmic membrane
Control of cell cycle progression progression from one phase to another is tightly
regulated
Failure to alternate S phase with mitosis could result in cells trying to divide before their DNA has been replicated
► cells like these must be eradicated or else would cause catastrophe
Meiosis REDUCTION DIVISION (meiosis I): where homologous chromosomes line up in pairs, and separate (2n to n) SYNAPSIS is the paring of two homologous chromosomes that allow a possible chromosomal crossover to occur

begins with a diploid cell that has proceeded through S phase (each chromosome is double stranded)
in prophase 1, homologous chromosomes, condense, and may exchange genetic material by crossing over at a chiasma
paired homologous chromosomes are lined up in the central plane of the spindle
Anaphase 1: homologous chromosomes separate and move to opposite poles, but remain double stranded MEIOSIS II: are similar to mitosis
in males, these cells differentiate into sperm cells
in females, the meiotic divisions are not accompanied by cytokinesis, with only one of the four daughter cells ending up as an egg. the other nuclei form short‐lived 'polar bodies' that are extruded from the cell or degrade
OTHER INFO Chromosomes are essentially inactive during
mitosis. ► Their mitotically functional sites are
kinetochores, which engage with spindle fibres
CENTROSOME is composed of two CENTRIOLES at right angles
ASTER is a centrosomes with a spindle star
L6 Molecules of Life Major elements: C, O, H, N Smaller amounts: P, S Ca, Na Mg, Cl, K Trace amounts: F, Si, V Cr, Mn, Fe, Ni, Co, Cu, Zn, Se, Mo, I
Compounds composed of carbon backbones are said to be organic molecules.
Backbone of the major macromolecules are made of carbon (valence of four), and offers many possibilities such as chains, branched chains, rings, double and triple bonds.
► These are the hydrocarbons, and various accessory groups can be attached.
Water Is the universal medium for living systems and their
chemical reactions
Cellular reactions are performed in solutions
Hydrogen‐bonding characteristics allow it to remain liquid over a wider range of temps
The partial ‐ve charge at the oxygen end and the partial +ve charge at the hydrogen end allows water molecules to form hydrogen bonds with each other and other polar molecules
► A universal solvent for polar molecules ► Repels non‐polar molecules
COHESION: water molecules sticking to one another ADHESION: water molecules sticking to other molecules
Cohesion and adhesion forces are responsible for capillary action, the ability of water to rise in tubes of a very narrow diameter
► Also causes water to move through minute soil particle spaces to plant roots and rise to great heights in tree trunks
Biomolecules ‐ Major Macromolecules
Assembled by condensation (dehydration) reactions between repeating monomers to form polymers
The reverse reaction is hydrolysis ► Occurs during the digestion of proteins,
polysaccharides and nucleic acids
Proteins Has amino acids as monomers
Two or more amino acids form peptide bonds
(covalent) between an amino group and a carboxyl group to produce a polypeptide chain
FUNCTION & STRUCTURE
Are the most functionally diverse biomolecule (depending on the protein folding)
► Enzymes, structural proteins, contractile proteins, hormones (signalling), transport proteins, defence proteins
Composed of linear chains of amino acids that never branch
There are 20 amino acids that form proteins ► Each with different R groups and different
properties ► Some R groups are hydrophilic (hydroxyl,
serine) or hydrophobic (methyl) or aromatic (ring in phenylalanine) exile others are charged, or neutral, or indifferent sizes
PRIMARY STRUCTURE: amino acid sequence SECONDARY STRUCTURE: shapes of chains due to the formation of electrostatic and hydrogen bonds between nearby amino acids (alpha‐helices and beta‐sheets) TERTIARY STRUCTURE: bending and folding (e.g. stabilised bridges) residues (ionic and hydrogen bonding) QUATERNARY STRUCTURE: association of two or more polypeptides

Globular proteins are compact, largely spherical and have surfaces that allow them to bind in a highly specific manner to other molecules
► Are usually soluble in water ► e.g. enzymes, haemoglobin
Fibrous protein show a range of structures to reflect their biological function ‐ generally physically tough
► keratins (horns, feathers, skin, hair nails) ► Silks ► collagens (connective tissue, bones,
cartilage, exoskeletons) ► Elastin (connective tissues of arteries, skin
and ligaments) ENZYMES: are biological catalysts that speed up biochemical reactions
Nucleic Acids Has nucleotides as monomers
► Composed of a nitrogenous base (pyrimidine: C,U,T; purine: G,A), a pentose monosaccharide (deoxyribose or ribose), and a phosphate group
► Can also form ATP (adenosine triphosphate)
DNA exists as two long chains running in opposite directions (complementary to each other), wound together in a double‐helix
► C pairs with G with three hydrogen bonds ► A pairs with T (DNA) or U (RNA) with two
hydrogen bonds ► DNA replication depends on the
complementary nature of the double helix
RNA is typically single‐stranded Can form complex structures by hydrogen bonding
with complementary bases elsewhere in the molecule
► Enabling RNA to fold back on itself and form stem‐loop structures
mRNA: is a complimentary copy of the genes encoded by nuclear DNA
► specifies the amino acid sequence of a given polypeptide
rRNA: a catalyst combines with ribosomal proteins to form ribosomes tRNA: molecules carry each of the 20 amino acids to the ribosomes for assembly into polypeptides in an order specified by the base sequence in mRNA Micro RNA: ~22 nucleotides long coded by DNA do regulate gene expression
► Do not code for proteins but control the RNAs that do
DNA
DNA is the blueprint for proteins with information stored in the "genetic code"
This code has been "cracked" and we can now "read" the recipe for life, we can also alter the code (genetic engineering) to create any recipe we choose
3 base system where a sequence of 3 bases code for 1 amino acid (e.g. ATG = methionine or TGG = tryptophane)
Information in gene is read off by transcription into messenger RNA
RNA is normally single stranded
mRNA leaves the nucleus and goes to a ribosome for translation into a chain of amino acids predetermined by the sequence of bases in the DNA of the gene
A polypeptide is assembled from individual amino acids at a ribosome according to the code carried by the mRNA from the gene
► DNA ‐> mRNA ‐> PROTEIN
Term Definition
Transcription The process of synthesising a single‐stranded RNA complementary to one of the strands or double‐stranded DNA
Primary transcript The initial transcript produced by RNA polymerase prior to RNA processing to produce the mature mRNA, rRNA or tRNA
mRNA The mature messenger RNA, containing one or more open reading frames that is translated by ribosomes to produce polypeptides
Intron Sequences that are removed during processing (maturation) of the primary mRNA
Exon Sequences that are joined together during processing to form the mature mRNA
Splicing The processing of a primary transcript to remove introns
5' and 3' untranslated sequences
Sequences in the mRNA ,located either side of the open reading frame, that do not encode a polypeptide
tRNA The adaptor RNA molecule that contains an anticodon complementary to a codon in the mRNA
Aminoacyl‐tRNA tRNA to which the appropriate amino acid has been covalently attached
Operon Sequences in bacteria DNA that encode a primary transcript and contain the cis‐regulatory sequences required for regulated expression of that transcript
Constitutive gene A gene expressed constantly
Translation The synthesis of a polypeptide by a ribosome directed by the sequence of codons in an mRNA

Open reading frame
A sequence of codons that begins with the AUG initiator codon, proceeds through a series of amino‐acid‐encoding codons and finishes with a termination codon
Ribosome The rRNA‐protein complex that provides a scaffold for the assembly of mRNA, peptidyl tRNA and aminoacyl tRNA and catalyses peptide bond formation during protein synthesis
Inducible gene A gene expressed only under certain conditions
Promoter The site to which RNA polymerase binds to initiate transcription
Operator A cis‐acting sequence to which a transcriptional repressor binds
Repressor A protein that binds to an operator and prevents transcription
Enhancer A eukaryotic cis‐acting regulatory sequence that controls expression of a gene, independent of its orientation or precise location with respect to the promoter for that gene
Transcription factor
A protein that binds to enhancer sequences and regulated transcription
DNA Replication The central Dogma (theory) of gene expression
asserts that amino acid sequence is not synthesised directly from DNA sequence, but in a two stage process.
► It states that DNA is transcribed into RNA which is then translated into protein.
TRANSCRIPTION: the first stage, synthesis of an RNA intermediate directly from a DNA template (a copy)
Occurs in the nucleolus ► Associated with regions of chromosomes
that have many tandemly repeated (head‐tail) copies of genes that encode rRNA
Binding RNA Polymerase to the promoter sequence of DNA to initiate synthesis and elongate the RNA chain
► Promoter only initiates transcription downstream from the promoter, which continues until it reaches a specific termination sequence on the DNA
► Then RNA polymerase ceases transcription and is released with the RNA product from the DNA template
In eukaryotes, most primary transcripts undergo a series of reactions that produce a modified mature RNA transcript
In bacteria, a single RNA transcribes all genes ► Ribosomes can bind and initiate translation
while transcription is still in process
► Only the most recently incorporated 10 nucleotides remain base paired to template, the rest peels away for translation
Chromatin is much less condensed to allow RNA polymerase to access DNA
Cutting, splicing and adding eukaryotic mRNA mRNA is modified before exporting from the
nucleus ► A modified guanosine triphosphate residue
is added to the 5' nucleotide to create a 'capped' RNA
► The 3' end of the primary transcript is cleaved and A residues added to create a tail of A residues (a poly A tail), termed polyadenylation
Pre‐RNA to mature RNA involved splicing separated segments, with removal of the sequences in between
► Genes include additional sequences that interrupt the sequences found in the mature mRNA
► Introns are removed ‐ removed by a large multi‐RNA and protein complex, the spliceosome
► Exons are kept TRANSLATION: RNA acts as a template for the synthesis of a polypeptide. A ribosome moves along the RNA, 'reading' the nucleotide sequence, and converts it into a specific amino acid sequence
A ribosome moves along an mRNA strand, translating codons into amino acids which are added to a growing polypeptide chain
► There are 64 possible combinations ‐ more than one codon codes for a specific amino acid
Reading Frames
Sequences at the 5' and 3' ends do not encode a protein
► 5' untranslated region (5' UTR) and 3' untranslated region (3' UTR)
In most cases an AUG codon serves as the start (initiator) codon
Stop codons: UAA, UAG, UGA

Open reading frames: the region of the mRNA that is read during the synthesis of a protein
Consists of a start codon followed by a series of codons that specify the sequence of amino acids and concludes with a termination
Reading frame mutations: different chemical mutagens cause mutations by altering DNA sequence during DNA replication.
Frameshift mutation: when one or two nucleotides are added or removed, all subsequent triplets is changed
► destroys the function of the gene and changes the amino acid sequence
Missense mutation: altering a codon to another amino acid
► leads to the production of an altered polypeptide
Nonsense mutation: change the sequence of a codon to a stop codon
► leads to premature termination of translation
Transfer RNA (tRNA)
Each type contains an anticodon sequence that covalently attaches to the complementary codon that specifies the amino acid to be attached to a polypeptide
► Brings the correct amino acid to the codon during protein synthesis
► The anticodon is found at the end of a loop in each tRNA molecule
Carbohydrates Polysaccharides are composed of polymers of
monosaccharides (sugars)
Main energy storage molecules
Structural component of plant cell walls
Sugars ‐ Monosaccharides / Disaccharides A sugar is a 3‐8 carbon molecule with a carbonyl
group C=O
e.g. glucose (aldehyde) [carbons numbered from ketone / aldehyde end]
The aldehyde group is reactive and links to the C5 hydroxyl group to form a ring ‐ the ring is not flat but liked like a deck chair
Fructose is a structural isomer of glucose, ketone form
A dehydration reaction between fructose and glucose produces a glycosidic bond resulting in a disaccharide
Polysaccharides Several saccharides linked together to form a
polysaccharide
Some common examples: ► Cellulose ‐ thousands of glucose, cell walls of
plants, most abundant organic molecule on earth
4 x 10^11 tonnes of carbohydrate per year from photosynthesis starch
Amylose and amylopectin with branches (sugars have several hydroxyl groups enabling branching)
Main storage molecule in plants ► Glycogen ‐ similar to amylopectin but more
branched, main storage molecule in animals ► Designer carbohydrates, including starch ‐ is
the latest technology
L8 Biological Membranes Lipids Fats and oils for energy storage and insulation
► Adipose tissue
Waxes for protection coatings ► Covers aquatic birds to make them
waterproof and buoyant ► Covers plants to prevent transpiration and
UV screening
Plants synthesise all their lipids
Chemical messengers and steroids
Structural components of membranes ► Karanoids ‐ vitamins
Characteristics of Lipids
Insoluble in water
Dissolve readily in organic solvents
Composed mainly of C, H and O
Differ from carbohydrates due to a smaller proportion of oxygen
► Good energy storage Lipids contain 37kJ/g of energy Proteins contain 17kJ/g of energy
May contain other elements ► Phosphorus and nitrogen
Common Lipids
Phospholipid Bilayer Membrane lipid
Physically separates contents of cells from extracellular / intercellular fluid, embedded with protein molecules (some span the bilayer)
Uncharged molecules can diffuse across the membrane along their own concentration gradient
Diffusion Diffusion of solutes in water Driven by the intrinsic kinetic energy of molecules

The passive net movement of molecules along their own concentration gradient, from a region of high concentration to a region of low concentration; requires no input of energy
► Spreads evenly ► The larger the concentration gradient, the
more rapid the rate of net diffusion ► Increasing the temperature also increase the
rate of diffusion
Electrochemical gradient The direction of passive movement of ions is
affected by the electrical gradient as well as the concentration gradient
► The difference in charge between the fluid compartments on either side
► These differences generate small voltage differences across membranes that affect the diffusion of ions
► +vely charged ions tend to move toward electrically ‐ve regions
► ‐vely charged ions will be repelled by other ‐ve charges
The electrical gradient may be the same direction as the concentration gradient, or they may be opposite
The direction of net passive movement will depend on the relative strengths of the electrical and concentration gradients
► The electrochemical gradient is the combination of these two
Equilibrium is rarely achieved due to factors (e.g. low membrane permeability)
► The action of transporter proteins and metabolic conversion of molecules is needed
Membrane Transporters Permeability for larger or charged molecules may
be too low to satisfy metabolic requirements
Membrane transporters (protein) accelerate the movement of molecules across membranes
► Solute specific ► Rate of transport depends on the availability
of these proteins At high concentrations, the rate may
level off due to completely occupied proteins
Transport is faster
Transport proteins become saturated as substrate concentration increases
Transport proteins are specific for substrates
Transport is inhibited by similar substrates that compete for the binding site
Facilitated Diffusion (passive protein‐mediated diffusion): the sue of transport proteins the accelerate the movement of a molecule down its concentration gradient
► Channels: act as pipes that allow direct passage through the membrane rapid
► Carriers: undergoes a conformational change when a solute binds to the side of the
membrane, this change moves the solute to the other side. The carrier reverts to its original state upon releasing the solute slow
Voltage‐gated channels open or close in response to changes in the voltage across the membrane
Ligand‐gated channels are activated by binding specific molecules (ligands)
Mechanically gated channels respond to physical disturbances
Aquaporin: a family of membrane‐spanning proteins that contribute to the water permeability of membranes (used by several other neutral solutes as well)
► Present in gall bladder, kidney, plant roots, etc.
Active Transport The use of metabolic energy to drive solutes against their electrochemical gradients
Primary active transport: the movement of the solute is directly linked to the hydrolysis of ATP via transport ATPase
► Pumps ions against their electrochemical gradient, ions move back across the membrane down their concentration gradient
Secondary active transport (co‐transport): A two‐stage process in which transport ATPase's are first used to generate an electrochemical gradient for an ion, whose downhill movement back across the membrane can be used to drive the uphill movement of another solute against its electrochemical gradient
Co‐transport: the coupling of the movements of two solutes
► Symport two solutes moving in the same direction
► Antiport: two solutes moving in opposite directions
► Although both molecules move across the membrane, only one is moving against its electrochemical gradient
Voltage Differences across membranes The Na+ ‐ K+ ATPase is responsible for maintaining
low intracellular Na+ ion and high intracellular K+ ion concentration

For each ATP hydrolysed, three Na+ ions are pumped out, only two K+ ions are pumped in
Net export of one +ve charge Membrane potential: the small voltage difference on both sides Electrogenic pumps: pumps that generate electrical gradients by pumping different amounts of charged ions in and out 1. By making the cytoplasm ‐ve, they provide an
electrical gradient for the passive uptake of cations 2. The pumping out of H+ or Na+ ions generates an
inward electrochemical gradient for these ions that can be used in the secondary active transport of other solutes
3. Changes in membrane potential can be used to collapse the voltage difference rapidly as ions flow down their electrochemical gradients
Osmosis Passive movement of water from a region of high water potential (low [K+]) to one of lower potential (high [K+])
Affected by solutes dissolved and physical pressure Water potential (Ψ): the overall free energy of water
The sum of the osmotic potential (Ψπ): accounts for the effect of solutes, and pressure potential (ΨP): which accounts fot the affect of pressure
Ψ = Ψπ + ΨP Isotonic (iso‐osmotic): concentration of solutes in surrounding water and in cells is equal Hypotonic (hypo‐osmotic): water potential in beaker is higher than in the cell, water diffuses in, cell swells
Turgid wall prevents haemolysis Hypertonic (hyperosmotic): water potential in beaker is reduced, red blood cell collapses Haemolysis: when a cell bursts from an excess of osmosis (irreversible) Plasmolysis: water diffuses out by osmosis and the cell collapses (when surrounding water potential is v.low) (plants only) Crenate: when a cell collapses when water flows out by osmosis (animals only)
Transport of large molecules Vesicle‐mediated transport
Endocytosis: a small area of plasma membrane enfolds (invaginates), enclosing substances that are outside the cell
► Phagocytosis: movement of solids
► Pinocytosis: movement of liquids
Receptor‐mediated endocytosis: endocytosis stimulated by initial binding of the solute to a receptor molecule ‐ very specific
► The internal surface is coated with the protein clathrin, which anchors and the receptor protein can causes the membrane to fold inwards to form a pit when activated
Exocytosis: intracellular vesicles fuse to the plasma membrane and the contents of the vesicle are deposited on the outside of the cell
L9 Respiration Metabolism: chemical and energy transformation in cells Metabolic Pathways: sequence of chemical reactions Potential energy is stored energy. Kinetic energy is energy expressed as movement
Enzymes Are biological catalysts that lower the activation
energy in substrates ► Catalysed reactions require less activation
energy
Enzyme Catalytic cycle
Electron transport pathways Enzymes for particular pathways are often
physically linked ‐ substrate channelling
Electrons transferred from donor to acceptor ► Molecule that loses electron is oxidised ► Molecule that gains electron is reduced
Transfer reactions are oxidation‐reduction reactions (redox)
Cellular Respiration Glycolysis